清华新闻网2月14日电 合成生物学的核心目标之一是通过设计并构建基因线路,精准控制生物体的行为,以实现特定的功能。在线路设计过程中,可预测设计是提高设计效率和实现精确控制的关键。可预测的基因线路设计使科学家能够更高效地调控生物体对外界刺激的响应,从而动态调控其表型。植物作为地球上最重要的生命形式之一,因其在农业和环境适应方面的巨大潜力,成为了基因线路设计和改造的重要研究对象。然而,植物系统的高度复杂性与较长的生长周期,使其在基因线路研究领域相对滞后。因此,开发一种高效、可预测的基因线路设计框架,对于加速植物合成生物学的发展和应用具有重要意义。
近日,清华大学药学院张数一团队提出了一种新型植物基因线路设计框架。该研究通过引入相对启动子单位(RPU)概念,结合原生质体瞬时表达系统和定量建模方法,实现了遗传元件的快速表征,大幅提高了研究效率,将实验迭代周期从大于2个月缩短到了10天之内。研究团队进一步开发了正交传感器和NOT门元件库,并基于精确的数学模型预测,成功设计并构建验证了可在植物中动态调控表型的基因线路。这一研究框架为植物基因线路的可预测设计和表型重编程提供了新方法,探索了从基因线路可预测设计到表型重编程的连接。
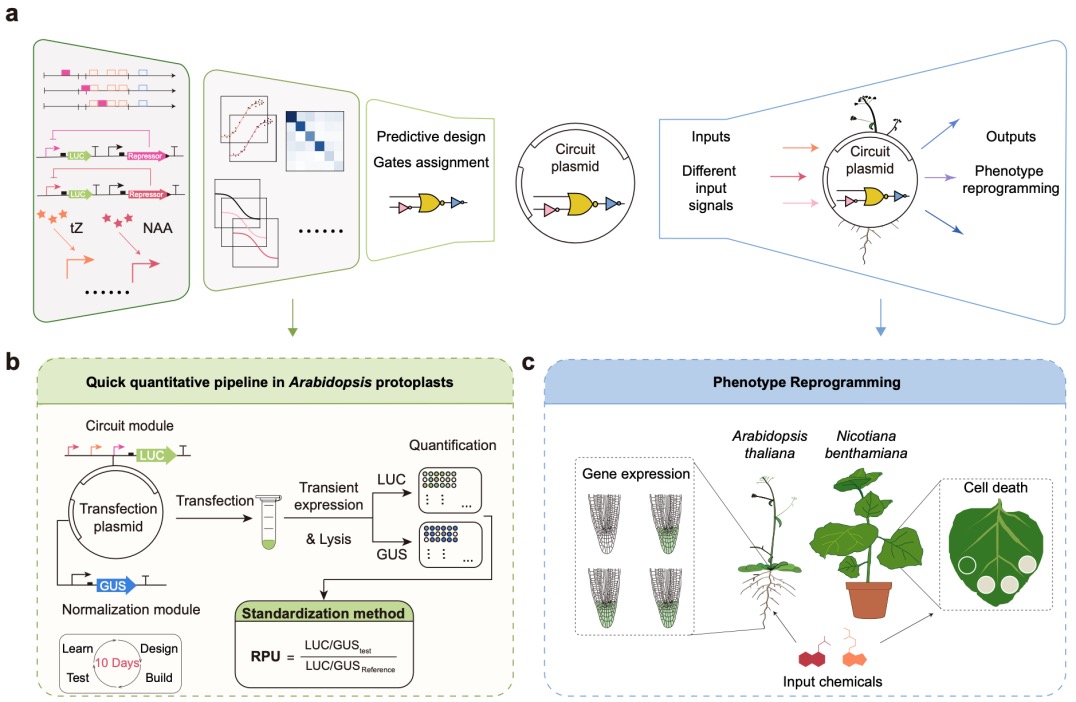
图1.植物基因线路可预测设计框架
在基因线路设计中,遗传元件的定量表征是确保设计可预测性的基础。然而,传统的植物系统表征方法耗时且易受实验条件变化的影响,难以实现高效和稳健的表征。为此,研究团队在本研究中引入了相对启动子单位(RPU, Relative Promoter Unit)的概念。RPU是一种基于标准化方法的定量指标,能够消除实验条件对启动子强度测量的影响。结合原生质体瞬时表达系统,研究团队成功建立了一个快速且准确的遗传元件表征平台。
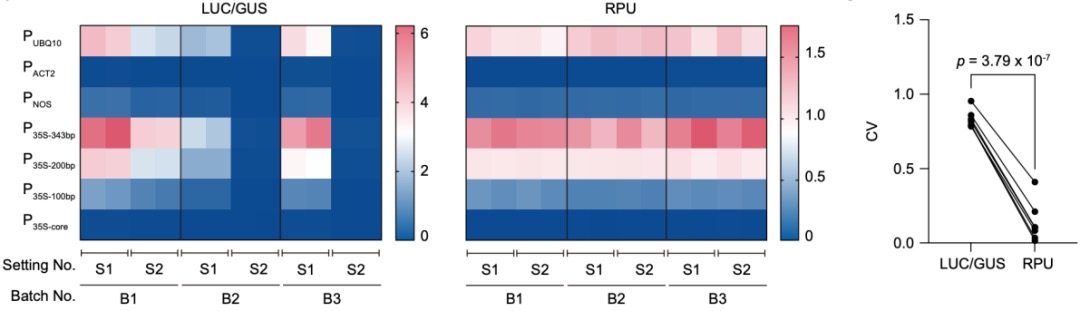
图2.RPU作为原生质体的标准化定量方法
在这一平台上,研究团队设计并表征了一组正交的遗传元件,包括不同强度的启动子和转录因子;随后测试表征了一组正交传感器与NOT门元件库,为基因线路组装和表型重编程奠定基础。在得到了经过良好表征的传感器与NOT门元件之后,研究团队成功构建并验证了21个基因线路,涵盖14种逻辑运算类型。更为重要的是,通过将元件参数纳入基因线路拓扑结构模型,研究团队实现了对基因线路性能的高度预测(R2=0.81)。
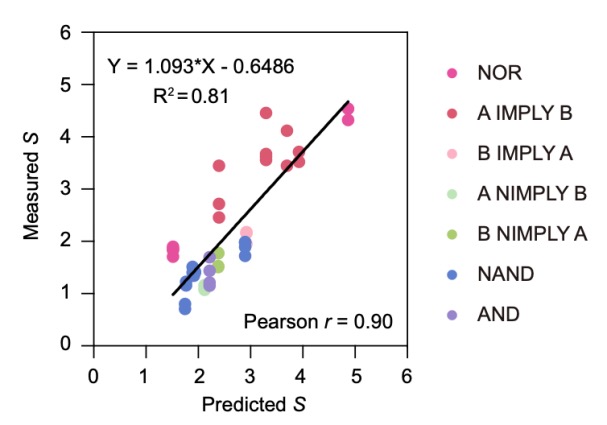
图3.可预测的基因线路设计
最后,研究团队验证了这一框架在活体植物中的适用性。分别以拟南芥和烟草为实验模型,设计并测试了能够实现多状态表型控制的基因线路。这些线路可根据输入化学信号的不同动态调控植物表型。在拟南芥中,通过基因线路设计精确调控了根部基因表达模式,实现了对外界信号的逻辑判断;在烟草中,应用基因线路成功实现了对超敏反应(HR, Hypersensitive Response)的逻辑控制。与目前植物中常见的硬编码基因线路(hard-coded)相比,这一设计框架表现出独特的优势。硬编码线路通常需要针对每种表型状态独立设计并转化特定的DNA序列,过程复杂且难以灵活调控。而新框架通过整合正交元件和精确建模,在单一线路中实现了多状态的控制,无需改造基因序列。
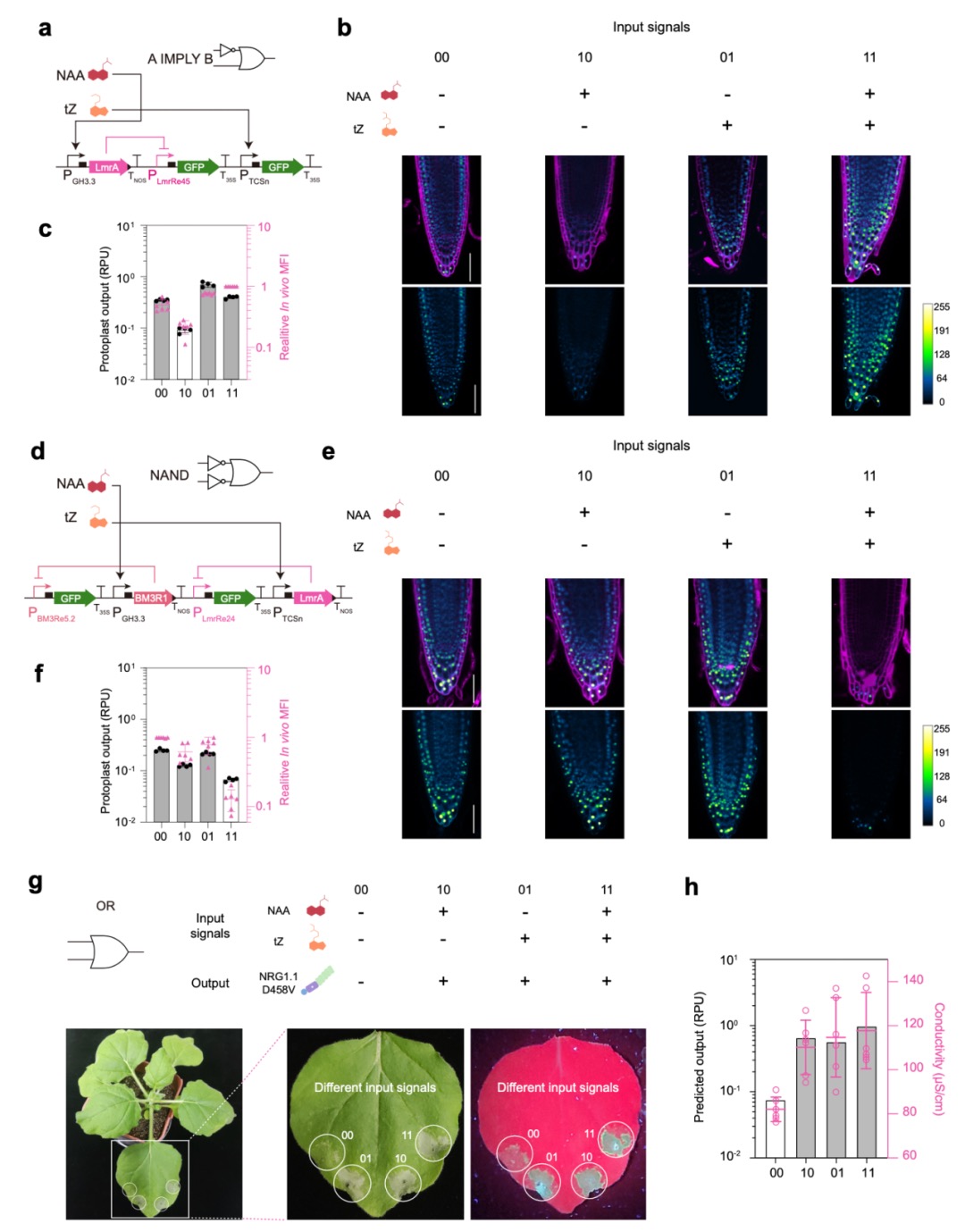
图4.可预测基因线路在活体植物中的表现
该研究提出的植物基因线路可预测设计框架为植物合成生物学开辟了新方向,加速了遗传元件表征和线路设计过程,同时在植物系统中实现了高效、可预测的表型重编程。这一框架的成功应用展示了植物基因线路可预测设计的潜力,为植物的基因工程和代谢优化等方面提供了更多可能。随着这一框架的进一步完善和应用,未来植物的基因改造将变得更加精准和高效,推动植物生物技术和绿色农业的持续发展。
相关研究成果以“植物表型重编程的预测遗传线路设计”(Predictive genetic circuit design for phenotype reprogramming in plants)为题,于1月16日发表于《自然·通讯》(Nature Communications)。
清华大学药学院已毕业博士孔辞为论文第一作者(现工作单位为北京生命科技研究院),清华大学药学院张数一副教授为论文通讯作者。清华大学生命学院博士生杨银和生命学院、植物生物学研究中心齐天从研究员为该研究作出了重要贡献。研究得到国家科技部重点研发计划、国家自然科学基金、清华大学笃实专项基金和北京生物结构前沿研究中心的资助。
论文链接:
https://www.nature.com/articles/s41467-025-56042-2
供稿:药学院
编辑:李华山
审核:郭玲